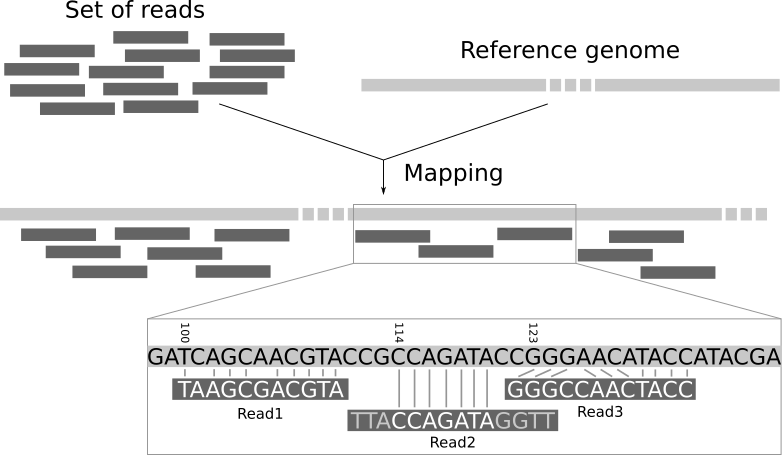
Los sistemas de secuenciación masiva han revolucionado muchos aspectos de la biología y la medicina personalizada ya que reducen el coste de obtención e incrementan la capacidad de producción de datos genómicos.
Estos datos producidos por los sistemas NGS (del inglés Next-generation sequencing) toman la forma de millones de fragmentos pequeños llamados "reads". En la gran mayoría de procedimientos de análisis de estos datos es necesario determinar la localización de cada read en el genoma de referencia.
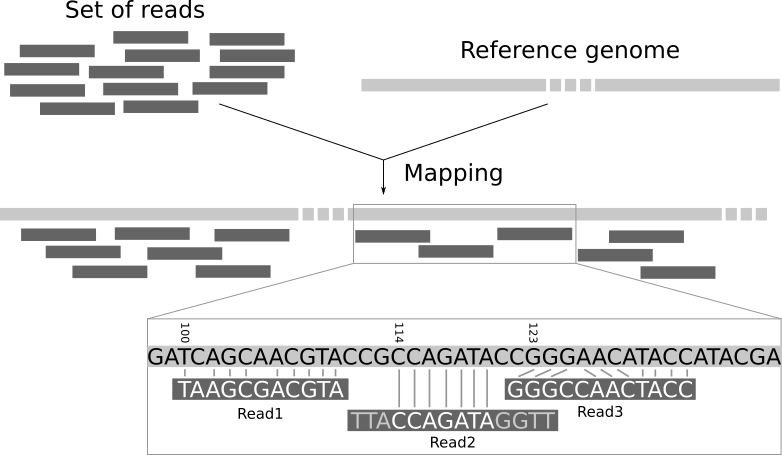
Este problema, llamado read-mapping o read-alignment se resuelve con herramientas específicas llamadas mappers. La utilización del nuevo algoritmo wavefront (WFA) [2] con complejidad proporcional al tamaño de la secuencia y al ratio de error entre secuencias permite realizar alineamientos más largos con una eficiencia superior. Este procedimiento es relevante de cara a procesar datos que provienen de las nuevas generaciones de sistemas de secuenciación donde se incrementa significativamente la longitud del read.
Los sistemas FPGA (del inglés field-programmable gate array) son dispositivos semiconductores que permiten la definición de su funcionalidad mediante su reconfiguración. Estos dispositivos se pueden personalizar para acelerar el procesamiento de algunas cargas de trabajo específicas con un coste energético muy reducido.
En el marco del proyecto DRAC, se ha realizado una implementación del algoritmo WFA basada en sistemas FPGA. Este acelerador FPGA está encargado de computar el alineamiento de pares de secuencias y de generar los resultados en una forma compacta que facilita la comunicación con la CPU del sistema. En una fase posterior, esta CPU se encarga de encontrar los resultados finales del alineamiento.
El diseño del acelerador se compone de múltiples elementos que colaboran en la computación de los alineamientos de secuencias. El diseño propuesto permite la adaptación a las características del conjunto de datos a procesar según el tamaño de las secuencias o reads y el ratio de error entre reads. Estos valores determinan los recursos requeridos para cada elemento alineador en el diseño de la FPGA. Por tanto, tendremos un determinado número de estos elementos a partir del número de recursos que disponga la plataforma FPGA de destino.
Los resultados de este estudio permiten comprobar aceleraciones en un sistema POWER9 entre 4,5x y 8,8x con una FPGA y entre 8,2x y 13,5x usando dos FPGAs. Del mismo modo, se reduce el consumo de energía entre un 6,1x y un 14,6x.
===
1: Joachim Wolff, Bérénice Batut, Helena Rasche, 2021 Mapping (Galaxy Training Materials). /training-material/topics/sequence-analysis/tutorials/mapping/tutorial.html Online; accessed Tue Jun 01 2021
2: Marco-Sola, S., Moure, J. C., Moreto, M., & Espinosa, A. (2021). Fast gap-affine pairwise alignment using the wavefront algorithm. Bioinformatics, 37(4), 456-463.
3: A. Hagui et al., An FPGA Accelerator of the Wavefront Algorithm for Genomics Pairwise Alignment. FPL 2021. International Conference on Field-Programmable Logic and Applications. 30 de Agosto al 3 de Setiembre. Technische Universität Dresden, Alemania.
