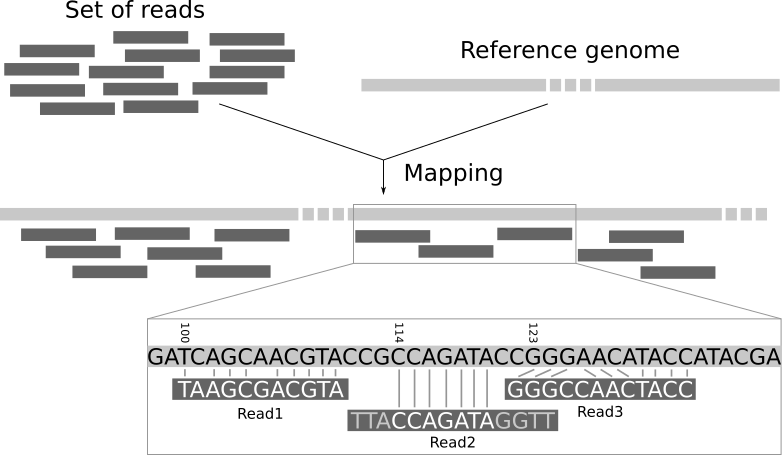
Els sistemes de seqüenciació massiva han revolucionat molts aspectes de la biologia i la medicina personalitzada ja que redueixen el cost d'obtenció i incrementen la capacitat de producció de dades genòmiques.
Aquestes dades produïdes per els sistemes NGS (de l’anglès Next-generation sequencing) prenen la forma de milions de fragments petits anomenats "reads". En la gran majoria de procediments d'anàlisi d'aquestes dades s'ha de determinar la localització de cada read en el genoma de referència.
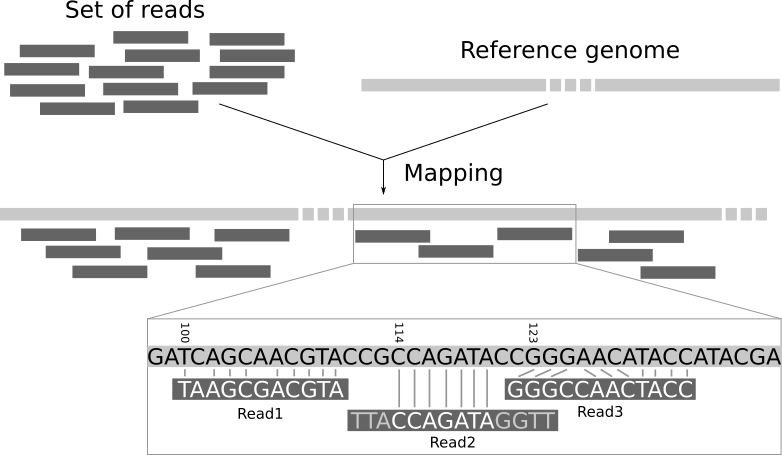
Aquest problema, anomenat read-mapping o read-alignment és resolt amb eines específiques anomenats mappers. La utilització del nou algorisme wavefront [2] amb complexitat proporcional a la longitud de la seqüència i la ràtio d'error permet fer alineaments mes llargs amb una millor eficiència. Rellevant per a les noves generacions de sistemes de seqüenciació on s'incrementa significativament la longitud del read.
Les FPGA (de l’anglès field-programmable gate array) son dispositius semiconductors que permeten la definició de la seva funcionalitat mitjançant la seva reconfiguració. Aquests dispositius es poden personalitzar per accelerar el processament d'algunes càrregues de treball específiques amb un cost energètic molt reduït.
En el marc del projecte DRAC, s'ha realitzat una implementació basada en sistemes FPGA de l'algorisme wavefront[3]. L'accelerador FPGA és l’encarregat de computar l'alineament de parells de seqüències i generar els resultats en una forma compacta que
facilita la comunicació amb la CPU del sistema. Després aquesta CPU s'encarrega de trobar els resultats finals.
El disseny de l'accelerador es composa de múltiples elements que col·laboren en la computació dels alineaments de seqüències. El disseny proposat permet l'adaptació a les característiques del conjunt de dades a processar segons la mida de les seqüencies o reads i la ràtio d'error entre els reads. Aquests valors determinen els recursos requerits per cada element alineador del disseny. Per tant tindrem un determinat número d'aquests elements a partir del número de recursos que tingui la plataforma FPGA de destí.
Els resultats d'aquest estudi permeten comprovar acceleracions en un sistema POWER9 entre 4,5x i 8,8x amb una FPGA i entre 8,2x i 13,5x amb dues FPGAs mentre es redueix el consum d'energia entre un 6,1x i un 14,6x
===
1: Joachim Wolff, Bérénice Batut, Helena Rasche, 2021 Mapping (Galaxy Training Materials). /training-material/topics/sequence-analysis/tutorials/mapping/tutorial.html Online; accessed Tue Jun 01 2021
2: Marco-Sola, S., Moure, J. C., Moreto, M., & Espinosa, A. (2021). Fast gap-affine pairwise alignment using the wavefront algorithm. Bioinformatics, 37(4), 456-463.
3: A. Hagui et al., An FPGA Accelerator of the Wavefront Algorithm for Genomics Pairwise Alignment. FPL 2021. International Conference on Field-Programmable Logic and Applications. 30 d’Agost al 3 de Setembre. Technische Universität Dresden, Alemanya.
