El alineamiento de secuencias es todavía hoy un problema fundamental con diversas aplicaciones prácticas como el reconocimiento de patrones, la seguridad de las redes de comunicación o la biología computacional.
Los métodos tradicionales se basan principalmente en técnicas de programación dinámica que requieren una gran cantidad de memoria principal y presentan muchas limitaciones para el procesamiento de datos de entrada de gran volumen.
En el marco del proyecto DRAC se ha realizado una implementación basada en sistemas GPU (Graphics Processing Unit) del algoritmo Wavefront [1] para calcular el alineamiento de secuencias genómicas utilizando la distancia exacta de edición.
La propuesta presentada [2] aprovecha las similitudes entre las dos secuencias de entrada para acelerar el proceso de alineamiento y reducir la cantidad de memoria que se necesita. Se presenta una representación de los datos de alineamiento que permite un buen encaje en la memoria rápida de los sistemas GPU.
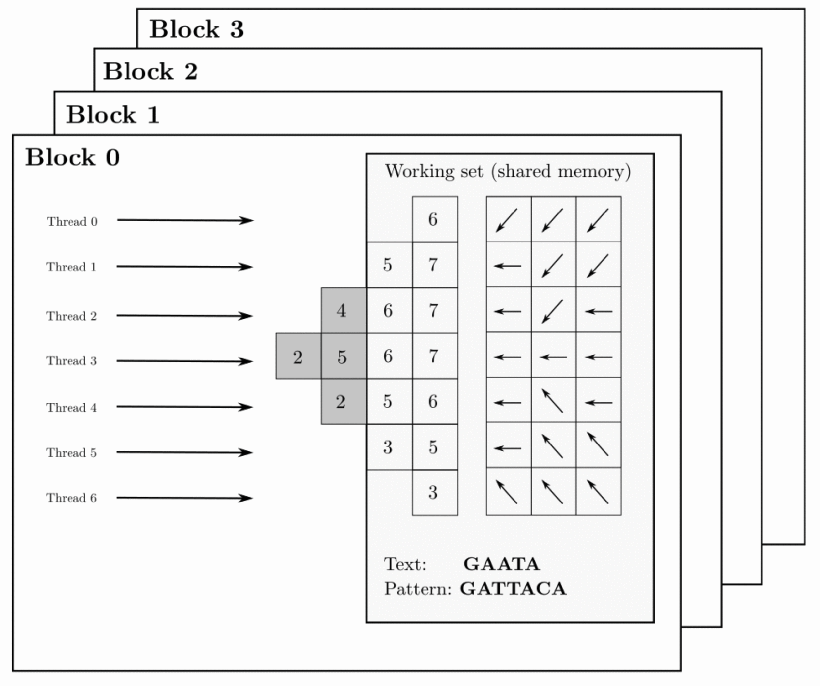
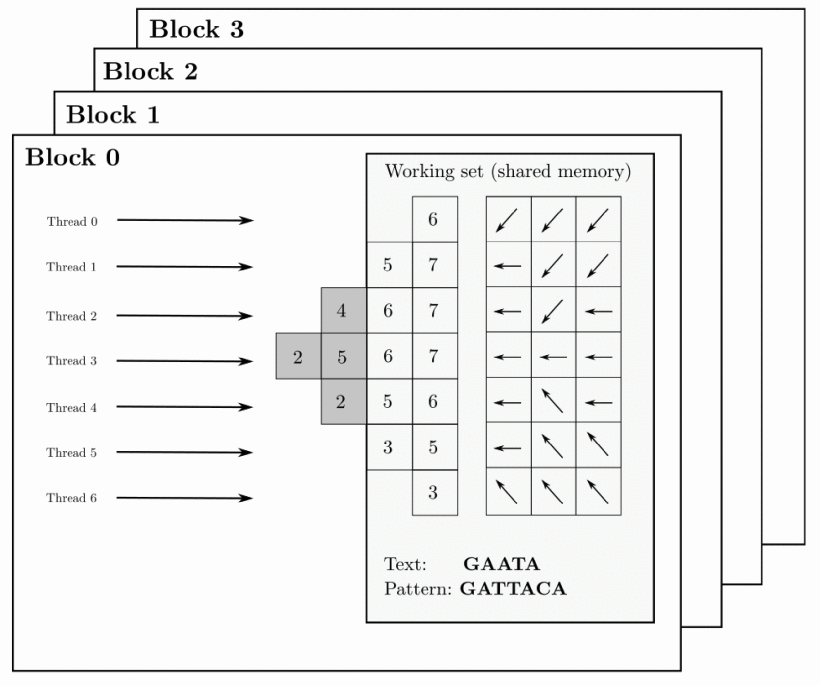
El análisis de grandes volúmenes de secuencias genómicas en los centros de datos requiere el alineamiento de millones de elementos de texto respecto a un genoma de referencia en un tiempo muy corto. Es necesario encontrar una estrategia que asigne un alineamiento a cada bloque de threads en ejecución en la GPU. De esta forma, todos los threads en un mismo bloque colaboran para realizar un alineamiento.
Esta aproximación reduce la necesidad de consumir memoria y registros de la GPU y potencia el acceso concurrente de diferentes bloques de ejecución a los datos como se muestra en la figura:
Los resultados de este estudio muestran una mejora entre 3 y 9 veces más rápida que la implementacion de WFA en un sistema multi-core con 20 núcleos. En un contexto más amplio, la propuesta WFA-GPU es 265 veces más rápica que las implementaciones actuales disponibles para CPU.
[1] Marco-Sola, S., Moure, J. C., Moreto, M., & Espinosa, A. (2021). Fast gap-affine pairwise alignment using the wavefront algorithm. Bioinformatics, 37(4), 456-463.
[2] Aguado-Puig, Q., Marco-Sola, S., Moure, J., Castells-Rufas, D., Alvarez, L., Espinosa, A., & Moreto, M. (2022). Accelerating Edit-Distance Sequence Alignment on GPU using the Wavefront Algorithm. IEEE access. 10, 63782-63796.
