L'alineament de seqüències és encara a dia d'avui un problema fonamental amb diverses aplicacions pràctiques com ara el reconeixement de patrons, la seguretat a les xarxes de comunicació o la biologia computacional.
Els mètodes tradicionals es basen principalment en tècniques de programació dinàmica que requereixen una gran quantitat de memòria principal i presenten moltes limitacions per al processament de dades d'entrada de gran volum.
En el marc del projecte DRAC, s'ha realitzat una implementació basada en sistemes GPU (Graphics Processing Unit) de l'algorisme Wavefront[1] per a calcular l'alineament de seqüències genòmiques utilitzant la distancia exacta d'edició.
La proposta presentada [2] aprofita les similituds entre les dues seqüències d'entrada per a accelerar el procés d'alineament alhora que redueix la quantitat de memòria que necessita. Es presenta una representació de les dades d'alineament que permet un bon encaix en la memòria ràpida dels sistemes GPU.
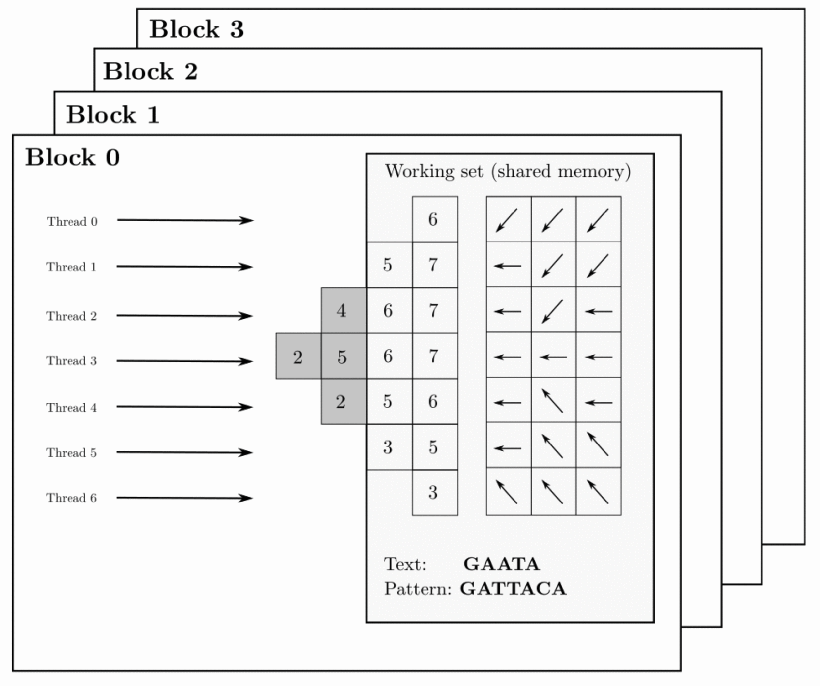
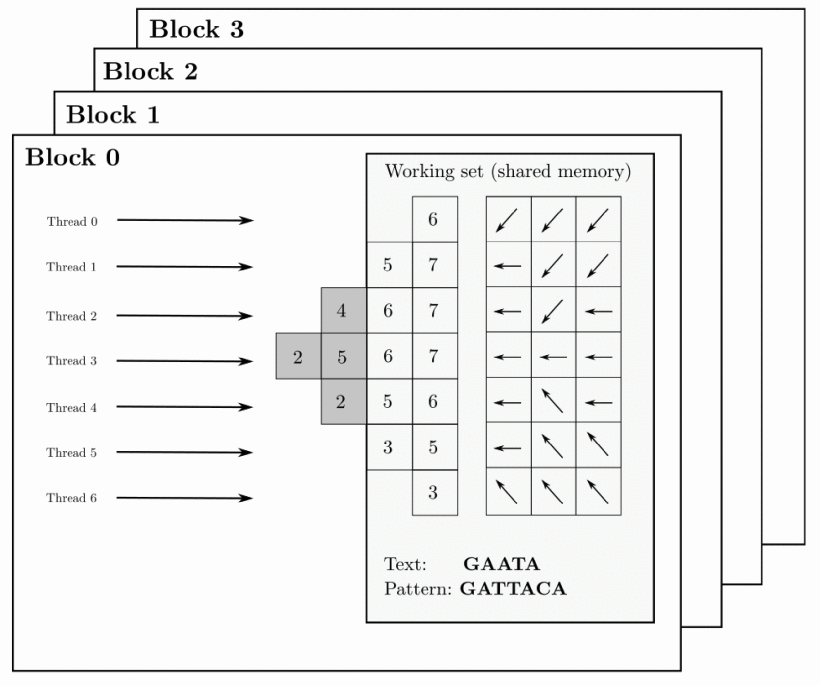
L'anàlisi de grans volums de seqüències als centres de dades requereix l'alineament de milions de elements de text respecte a un genoma de referència en un temps molt curt. És necessari trobar una estratègia que assigni un alineament a cada bloc de fils en execució a la GPU. D'aquesta forma, tots els fils en el bloc col·laboren per realitzar un alineament.
Aquesta aproximació redueix la necessitat de memòria i registres i potencia l'accés concurrent de diferents blocs d'execució a les dades com es mostra a la figura:
Els resultats d'aquest estudi mostren una millora entre 3 i 9 vegades més ràpid que la implementació de WFA en un sistema CPU multi-core amb 20 nuclis. En un context més ampli, la proposta WFA-GPU és 265 vegades més ràpida que les implementacions actuals disponibles per a CPU.
[1] Marco-Sola, S., Moure, J. C., Moreto, M., & Espinosa, A. (2021). Fast gap-affine pairwise alignment using the wavefront algorithm. Bioinformatics, 37(4), 456-463.
[2] Aguado-Puig, Q., Marco-Sola, S., Moure, J., Castells-Rufas, D., Alvarez, L., Espinosa, A., & Moreto, M. (2022). Accelerating Edit-Distance Sequence Alignment on GPU using the Wavefront Algorithm. IEEE access. 10, 63782-63796.
